
Back Ploïdie Afrikaans ቀመርነት Amharic صيغة صبغية Arabic Плоіднасць Byelorussian Хаплоиден набор Bulgarian ডিপ্লয়েড Bengali/Bangla Ploidija BS Ploidie Czech Ploidi Danish Ploidiegrad German

Ploidia é o termo designado para definir o conjunto numérico completo de cromossomas de uma determinada célula.
A depender da aplicação pode ser utilizado para comparações entre o conjunto numérico cromossômico tido como natural (normal) de determinado organismo vivo e conjuntos numéricos derivados, ou mutantes (desvios).
A ploidia pode ser também útil na comparação numérica de cromossomos entre células de organismos distintos, ou manifestações genômicas de um mesmo organismo que variam conforme o seu estágio de desenvolvimento.
Em organismos considerados mais complexos, células de diferentes tecidos ou conjuntos celulares, costumam variar em ploidia no mesmo indivíduo. Um bom exemplo disso é comumente observado ao distinguir células somáticas de células sexuais, ou gametas, dos mais variados grupos de seres multicelulares.
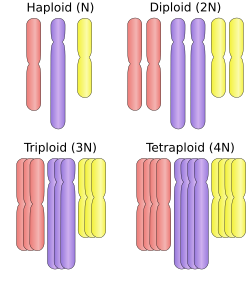
As formas mais frequentes derivadas de ploidia são: haploidia, diploidia e poliploidia.
Como ploidia refere-se a organizações numéricas de cromossomos, é um termo geralmente empregado apenas para tratar de genomas de eucariotos. Embora o termo haploide possa ser utilizado para designar o genoma bacteriano e viral, por possuírem uma estrutura de acoplamento genético único na maioria dos casos (DNA circular ou RNA circular em alguns vírus).
Podemos perceber, por exemplo, que o cariótipo comum de um Homo sapiens (2n = 2x = 22 pares de autossomos mais XX ou XY) e de uma fêmea de Characidium gomesi, um tipo de peixe triplóide (2n = 3x = 24 trios de autossomos mais ZZW) encontrado na Serra da Mantiqueira, Brasil [2], diferem-se profundamente quanto à ploidia.
Ploidia também refere-se portanto ao número de alelos possíveis para o conjunto de genes autossómicos e pseudo-autossómicos nela existentes. Células somáticas, tecidos e organismos indivíduais podem ser descritos de acordo com o seu nível de ploidia, ou seja do número de conjuntos de cromossomas homólogos presentes, como monoploide ou haploide (1 conjunto), diploide (2 conjuntos), triploide (3 conjuntos), tetraploide (4 conjuntos), pentaploide (5 conjuntos), e assim por diante.[3][4] O termo genérico poliploide é frequentemente utilizado para descrever situações em que estejam presentes três ou mais conjuntos de cromossomas.[5][6]
- ↑ Daniel Hartl (2011). Essential Genetics: A Genomics Perspective. [S.l.]: Jones & Bartlett Learning. 177 páginas. ISBN 978-0-7637-7364-9
- ↑ CENTOFANTE, Liano; BERTOLLO, Luiz A. C; MOREIRA-FILHO, Orlando. Comparative cytogenetics among sympatric species of Characidium (Pisces, Characiformes). Diversity analysis with the description of a ZW sex chromosome system and natural triploidy.ResearchGate, 2021. DOI:10.1080/00087114.2001.10589233
- ↑ U. R. Murty (1973). «Morphology of pachytene chromosomes and its bearing on the nature of polyploidy in the cytological races of Apluda mutica L.». Genetica. 44 (2): 234–243. doi:10.1007/bf00119108
- ↑ Tuguo Tateoka (maio 1975). «A contribution to the taxonomy of the Agrostis mertensii-flaccida complex (Poaceae) in Japan». Journal of Plant Research. 88 (2): 65–87. doi:10.1007/bf02491243
- ↑ Rieger, R.; Michaelis, A.; Green, M.M. (1976). Glossary of Genetics and Cytogenetics: Classical and Molecular 4th ed. Berlin/Heidelberg: Springer-Verlag. p. 434. ISBN 978-3-540-07668-1. doi:10.1007/978-3-642-96327-8
- ↑ Darlington, C. D. (Cyril Dean) (1937). Recent advances in cytology. Philadelphia: P. Blakiston's son & co. p. 60